エネルギー極小化計算とMD計算
GROMACSを使うまで
Windows版は、Windows 11 および Windows 10 ではユーザは GROMACS のインストールなしで GROMACS を使えます。
Linux版とMAC版は、ユーザが GROMACS をインストールします。[Preference] で、インストールした GROMACS の share と bin が存在する directory を設定してください。
※ MAC と Linux へのGROMACSのインストール方法はこちら。
※ 現バージョンでは、GROMACS の実行は、bin/gmx で、並列数の指定なしで行います。インストールディレクトリに、bin/gmx が存在しない時は実行できません。OpenMP や thread MPI の並列数は、GROMACS の自動的に並列数を指定する機能に依存してます。MPI は使用しません。
エネルギー極小化計算
基本的には、myPresto の cosgene によるエネルギー極小化計算の場合と同様に操作します。ただし、以下の制限があります。
- [Solvate] で作成する水溶媒の形状は、[Cube] だけが有効です。[Sphere] で作成した系は計算できません。周期的境界条件で計算するためです。
- PME法を使用してます。GUI からは、nsteps, integrator, define のみを修正でき、その他はデフォルト値で固定です。
MD計算
基本的には、myPresto の cosgene の MD計算の場合と同様に操作します。ただし、現バージョンでは GUI から設定できる項目に限りがあり、以下の制限があります。
- NVT アンサンブル、または、NPT アンサンブルの場合は PME法を使用してます。GUI からは、define, nsteps, dt, nstxout, nstvout, nstenergy, nslog, nstxout-compressed, continuation, gen_vel, gen_temp, tcouple, ref_t, pcouple, rvdw, rcoulomb のみを修正でき、その他はデフォルト値で固定です。
トラジェクトリ解析
以下のトラジェクトリ解析を実行して、時間軸のあるグラフは動画と連動したグラフを表示します。
一般
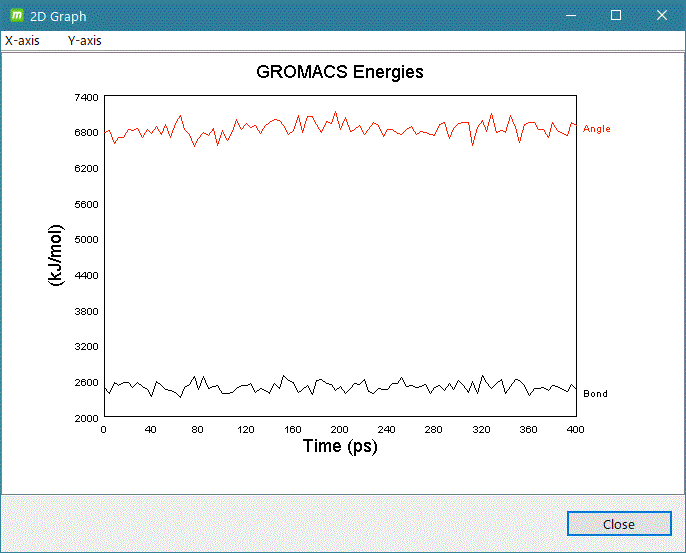
- gmx energy
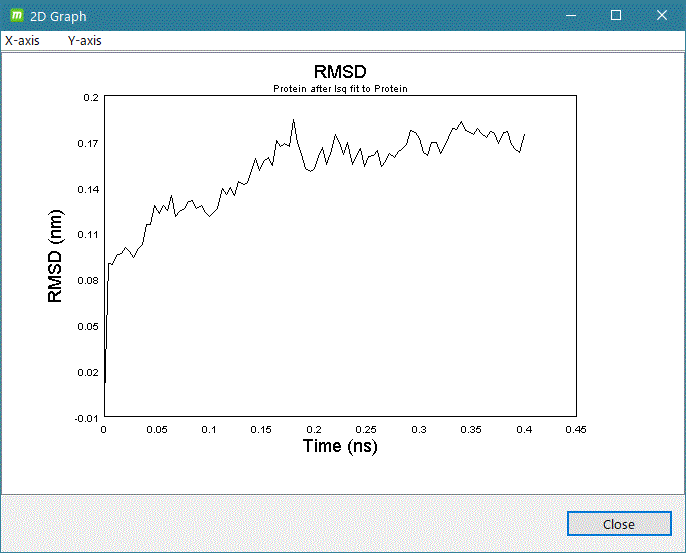
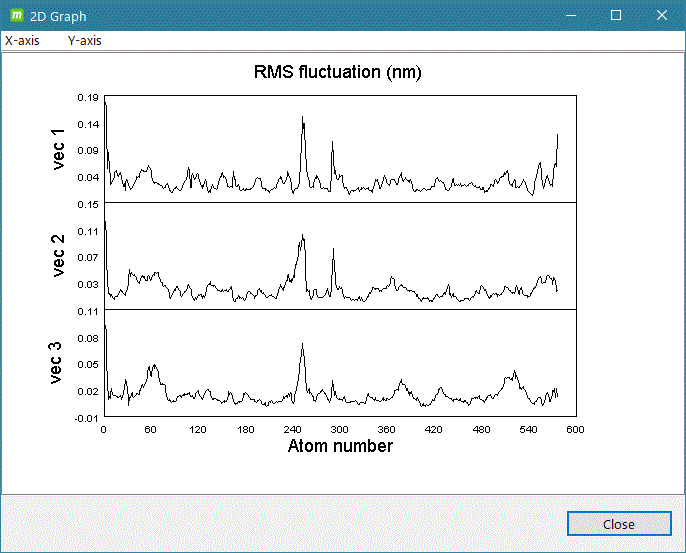
- gmx rms
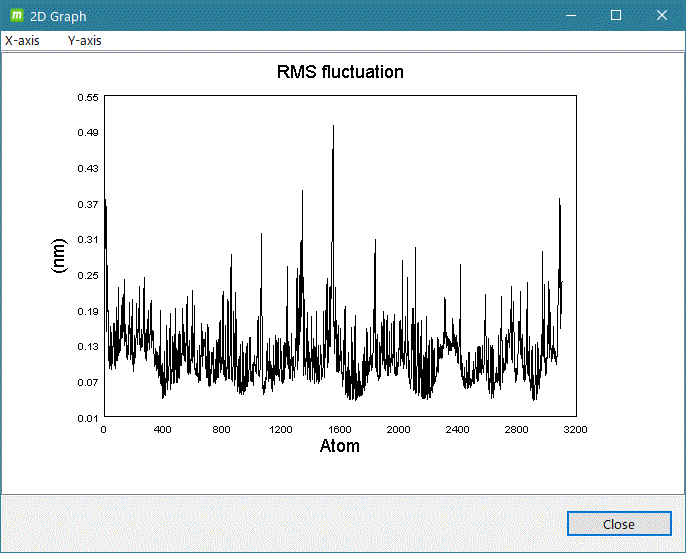
- gmx rmsf
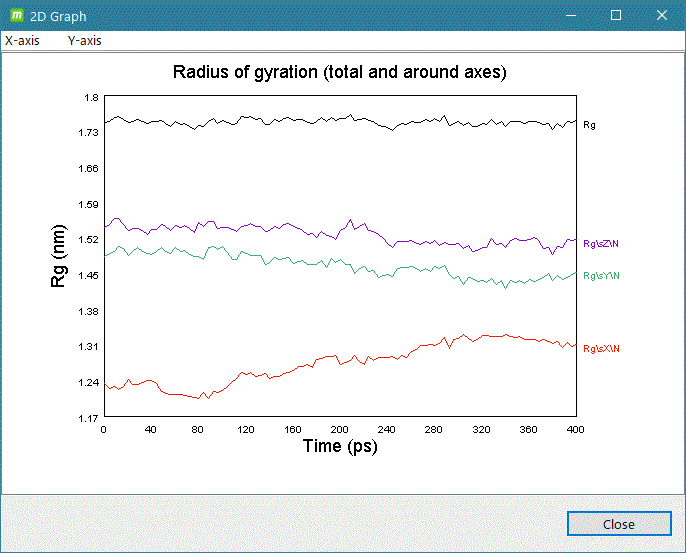
- gmx gyrate
- gmx mindist
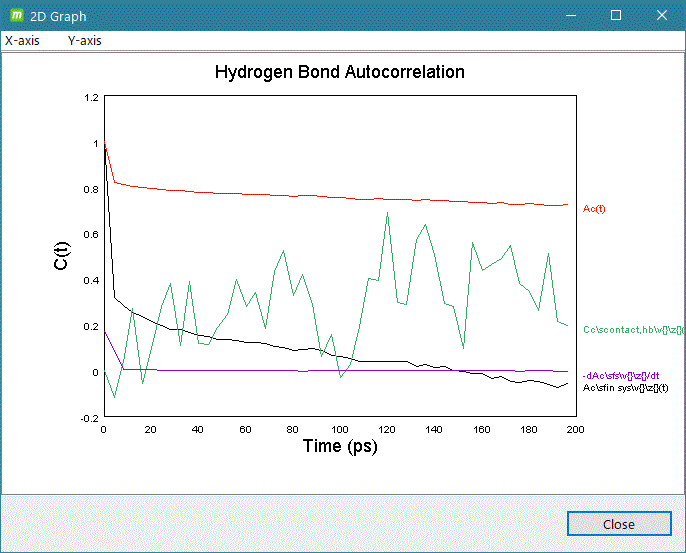
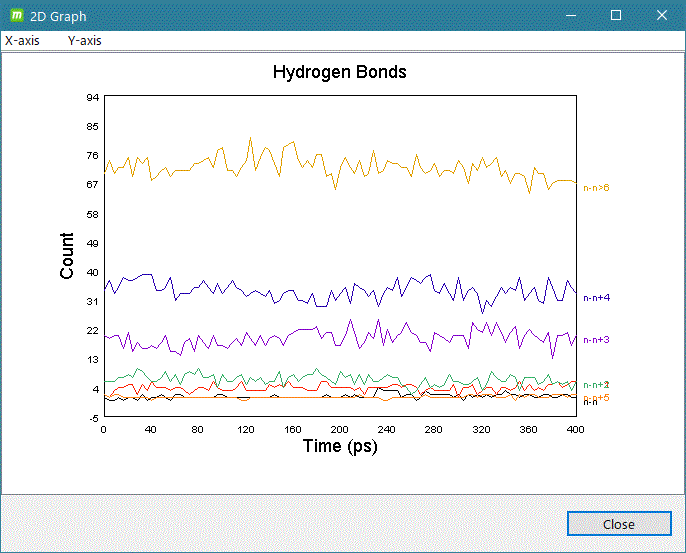
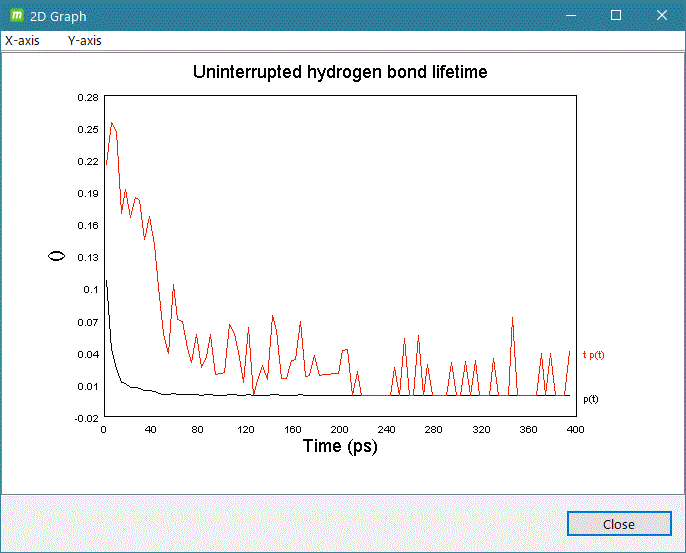
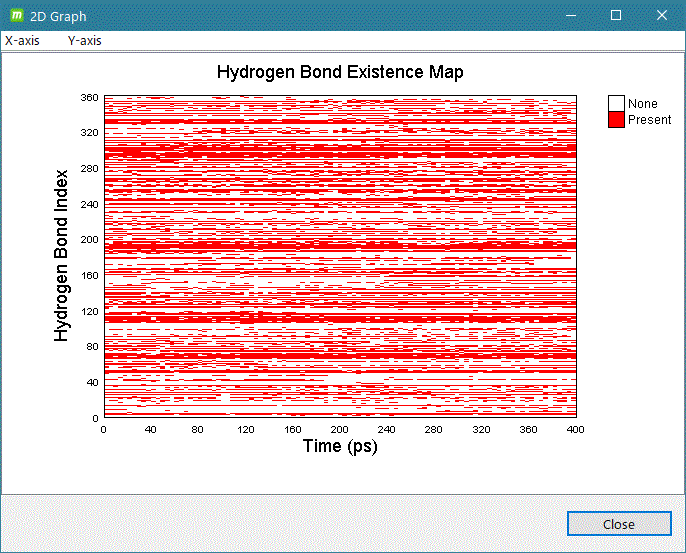
- gmx hbond
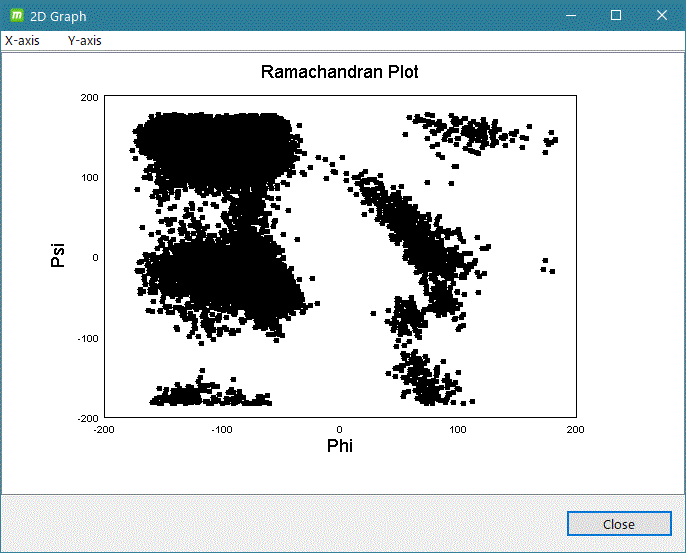
- gmx rama
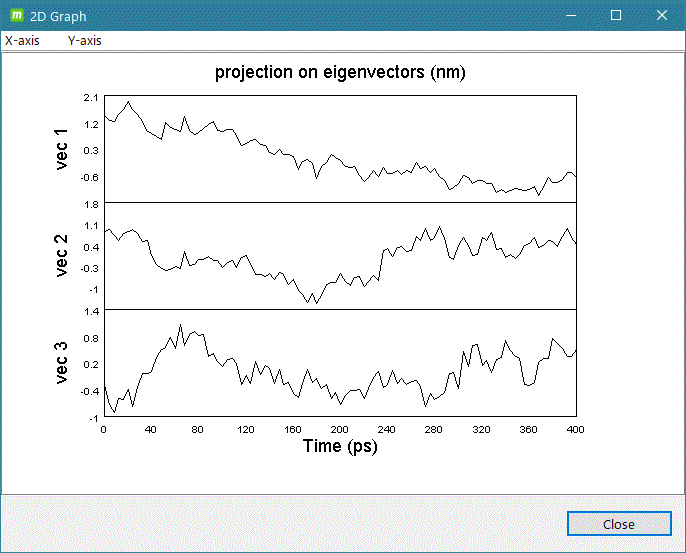
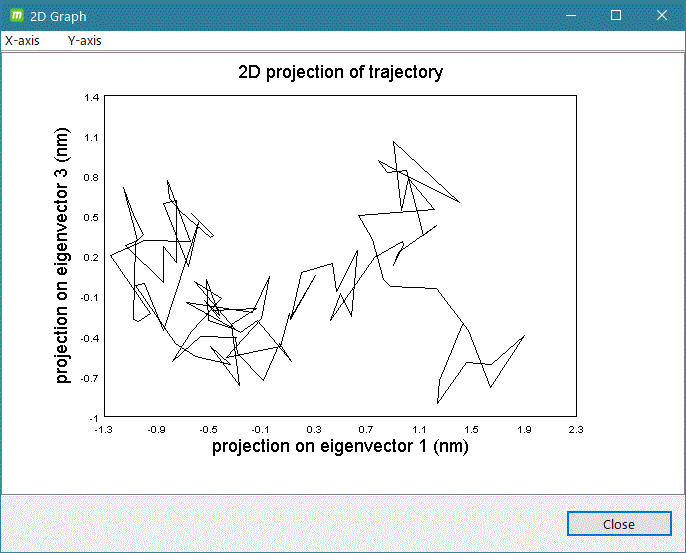
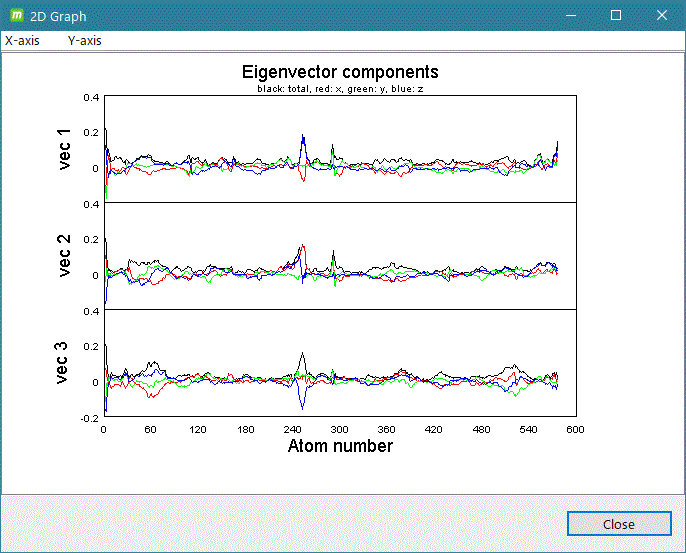
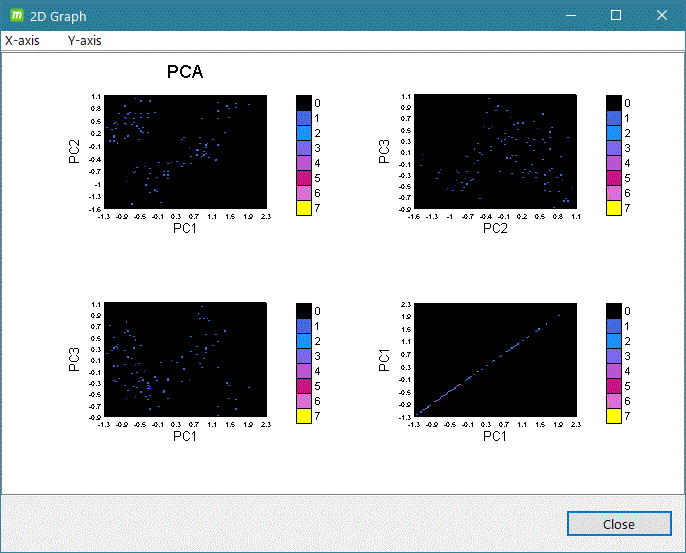
共分散解析 (Covarience analysis, PCA関係)
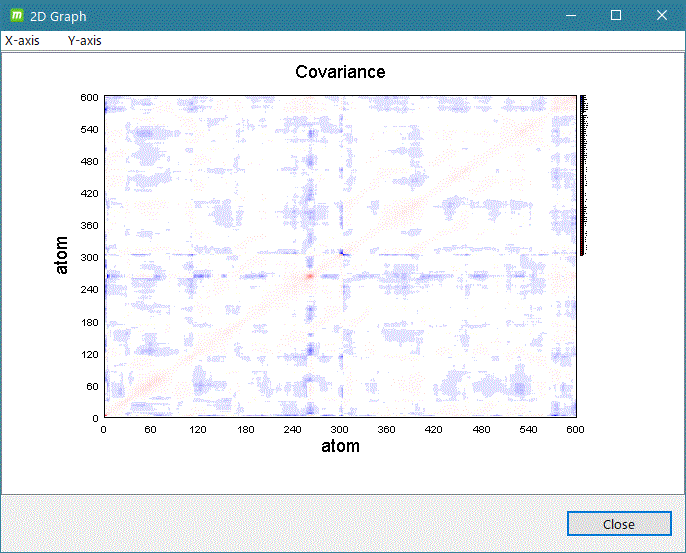
- gmx covar
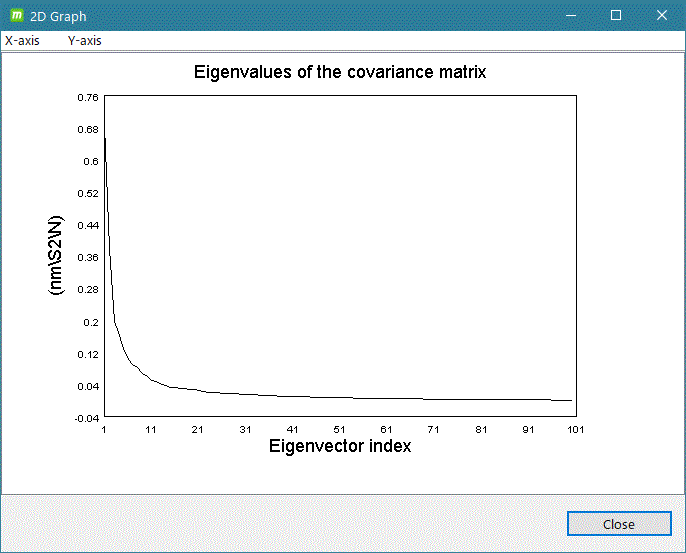
- gmx anaeig
※ 使用法の詳細は、「MolDesk Basicマニュアル」をご参照ください。
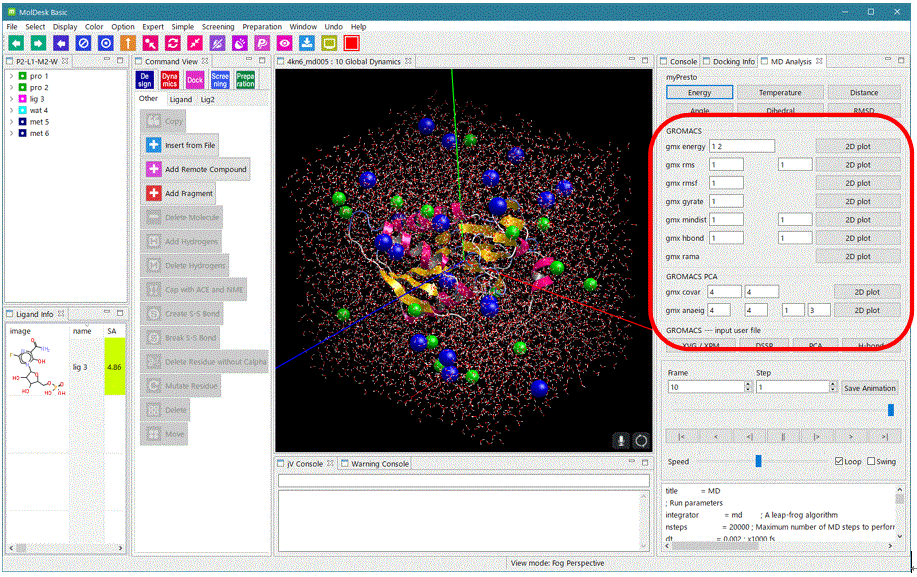
計算系の解析したいグループを入力して [2D plot] をクリックするとグラフ表示します。
グラフ表示例)